Echantillonnage basé sur le risque
L’échantillonnage est la sélection d’une partie d’un tout, typiquement une partie des individus d’une population, partie qui est ensuite étudiée afin de caractériser le tout. Il existe de nombreuses stratégies d’échantillonnage adaptées aux divers domaines d’application. Dans les domaines concernant la Plateforme ESV, on rencontre classiquement :
- les stratégies basées sur un tirage uniforme aléatoire des individus, tirage qui est éventuellement stratifié, i.e. qui repose sur des tirages uniformes aléatoires mais d’intensités différentes, dans des strates différentes de la population ;
- les stratégies basées sur un échantillonnage systématique dans lequel la sélection des individus est effectuée selon une grille régulière ;
- les stratégies basées sur le risque dans lesquels les individus sont tirés aléatoirement dans la population mais la probabilité de tirage de chaque individu dépend de son niveau ou de sa catégorie de risque.
L’échantillonnage basé sur le risque est particulièrement pertinent dans le cadre des stratégies de surveillance et de contrôle des ravageurs et des agents pathogènes. Le risque d’introduction ou d’invasion, le risque de développement, le risque de propagation ou encore le risque de dégâts environnementaux ou de pertes économiques sont autant de critères qui peuvent amener à prioriser la surveillance d’individus ou de sites ciblés. L’échantillonnage basé sur le risque est préconisé notamment par la Commission Européenne pour faciliter la détection précoce et pour des raisons d’efficacité et de coût.
RiBESS | Outil de calibration de la taille d'échantillonnage
► Description
RiBESS est un outil statistique en ligne développé par l’EFSA (European Food Security Safety). Cet outil fournit à l’utilisateur l'une des trois variables suivantes conditionnellement aux deux autres :
- la taille de l’échantillon requis pour la surveillance d’une zone (région/département, zone infectée ...) ;
- la sensibilité de l’échantillonnage (performance de l’échantillonnage) ;
- la probabilité que le territoire soit indemne d’une maladie en fonction du nombre d’échantillons réalisés.
► Exemple d'application
L’outil RiBESS a été testé dans le cadre de la surveillance de Xylella fastidiosa dans le sud-est de la France afin de donner des indications relatives au nombre et aux types de sites à inspecter. L’utilisation de RiBESS nécessite de renseigner diverses variables dont la taille de la population d’étude (i.e. le nombre d’unités épidémiologiques recouvrant la zone d’étude ; typiquement des quadrats d’une surface donnée), la sensibilité du test de diagnostic de la maladie (en l’occurrence le test PCR mis en œuvre par les laboratoires agréés pour Xylella fastidiosa), la prévalence maximale attendue, le taux de détection de la maladie dès que 1% de la population est infectée et le taux d’erreur que l’on est prêt à accepter (typiquement 5%).
Les unités épidémiologiques sont caractérisées par des variables explicatives du risque (ou facteurs de risque) telles que la présence ou la quantité de plantes hôtes dans l’unité, ou encore des variables environnementales connues comme étant reliées au risque de présence de l’organisme nuisible (e.g., un indicateur des températures hivernales minimales pour Xylella fastidiosa). RiBESS évalue alors automatiquement le nombre d’échantillons à collecter en fonction des risques relatifs calculés pour chaque facteur de risque.
► Notes et références
Ciubotaru, RM. et al. (2018). Technical report of the methodology and work-plan for developing plant pest survey guidelines. EFSA supporting publication 2018:EN-1399. 36pp.
EFSA (2012). A framework to substantiate absence of disease: the risk based estimate of system sensitivity tool (RiBESS) using data collated according to the EFSA Standard Sample Description - An example on Echinococcus multilocularis.
Spatialisation et optimisation de l’échantillonnage
► Description
En schématisant, la construction d’un plan d’échantillonnage dans un cadre spatial consiste à déterminer la répartition dans l’espace des échantillons ainsi que le nombre d’échantillons à collecter. Concernant la répartition spatiale des échantillons, diverses méthodes existent : répartition aléatoire pondérée par le risque, sélection des n sites les plus à risque, identification d’un ensemble de sites décentralisé avec la plus grande capacité d’infection (e.g., via un algorithme tel que VoteRank), identification d’un ensemble de sites qui sont les plus à même de disperser la maladie (e.g., via un algorithme tel que GRWA). Déterminer le nombre d’échantillons à collecter est un compromis entre coût (humain, financier, environnemental) et efficacité (mesurée par exemple par la capacité à délimiter précisément les zones infectées, ou la capacité à détecter de manière précoce les nouveaux foyers). Le calcul du gain marginal d’une stratégie d’échantillonnage (gain obtenu en ajoutant un échantillon) peut être représenté graphiquement par une courbe de l’efficacité en fonction du nombre d’échantillons. “L’optimum” de la courbe (i.e., le point au delà duquel le gain d’efficacité apporté par un échantillon supplémentaire est plus faible que le coût induit par cet échantillon supplémentaire) permet d’identifier la taille d’échantillonnage optimale. Cet optimum dépend en général du choix de la stratégie de répartition spatiale des échantillons.
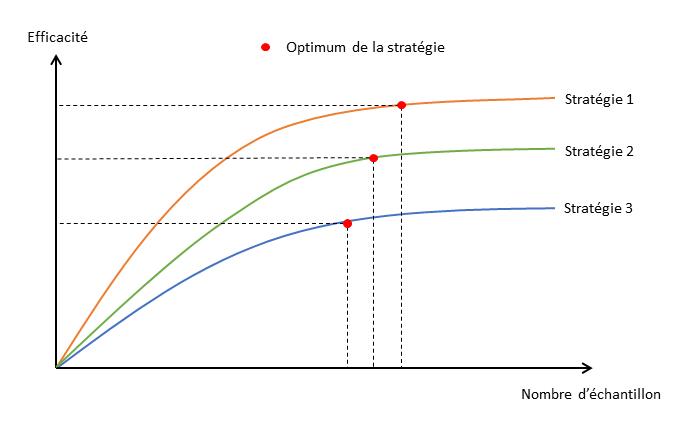
► Exemple d'application
Une approche de spatialisation de l’échantillonnage et de mise en concurrence de différentes stratégies a été mise en œuvre dans le cadre de la surveillance de Xylella fastidiosa en France continentale. Différentes stratégies basées sur des modulations diverses du risque et des tailles d’échantillons variables ont été comparées en fonction de leurs capacités à détecter de manière précoce l’émergence de la bactérie. Voir le poster ci-dessous.
► Notes et références
De Aruda, G. F. et al. (2014). Role of centrality for the identification of influential spreaders in complex networks. Physical Review E 90(3).
Marjou, M. et al. (2019). Risk-based surveillance strategies for early detection of Xylella fastidiosa in continental France. Poster. 2ème conférence sur Xylella fastidiosa. Ajaccio, octobre 2019.
Martinetti, D., & Soubeyrand, S. (2019). Identifying lookouts for epidemio-surveillance: application to the emergence of Xylella fastidiosa in France. Phytopathology, 109(2), 265-276.
Zhang, J.-X. et al. (2016). Identifying a set of influential spreaders in complex networks. Scientific Reports 6.

